| Numéro |
Cah. Myol.
Numéro 15, Juin 2017
|
|
|---|---|---|
| Page(s) | 17 - 21 | |
| Section | Mise au point / Focus | |
| DOI | https://doi.org/10.1051/myolog/201715017 | |
| Publié en ligne | 23 juin 2017 | |
Quel avenir pour la dystrophine ?
What future for dystrophin?
1
PhyMedExp, Université de Montpellier, Inserm U1046, CNRS, UMR 9214, 34295 Montpellier Cedex 5, France
2
Département de Neuropédiatrie - CR Maladies Neuromusculaires, hôpital Gui-de-Chauliac, 80, avenue Augustin-Fliche, 34295 Montpellier Cedex 5, France
3
PhyMedExp, Université de Montpellier, Inserm U1046, CNRS, UMR 9214, 34295 Montpellier Cedex 5, France
* Contact domimornet@gmail.com
En 1868, Duchenne de Boulogne décrivait cliniquement une maladie évolutive responsable d’une dystrophie musculaire qui porte aujourd’hui son nom, sans pour autant en comprendre l’origine. Il y a maintenant 30 ans des travaux de recherches menés par une équipe dirigée par le professeur LM. Kunkel (Boston, USA) sur cette dystrophie musculaire permettait d’identifier le gène DMD. La protéine résultante était alors baptisée : la dystrophine. Depuis les revues de mise à jour des connaissances acquises ont été très nombreuses sur ce sujet. Où en est-on en 2017 ? Cet article propose de faire le point sur la dystrophine et son environnement musculaire, les pathologies qui résultent d’une altération directe et/ou indirecte de la dystrophine ainsi que sur les perspectives expérimentales du traitement d’un déficit en dystrophine.
© D. Mornet et F. Rivier, publié par EDP Sciences, 2017
 Cet article est distribué sous licence « Creative Commons » : http://creativecommons.org/licenses/by/4.0/deed.fr/, permettant une ré-utilisation du contenu sans restriction à condition de mentionner clairement la source.
Cet article est distribué sous licence « Creative Commons » : http://creativecommons.org/licenses/by/4.0/deed.fr/, permettant une ré-utilisation du contenu sans restriction à condition de mentionner clairement la source.
La dystrophine et son environnement musculaire
Dès 1987, date de sa découverte [1], et durant environ 2 décennies la dystrophine voyait presque chaque année autour d’elle apparaître de nouveaux partenaires aussi bien ancrés dans la membrane musculaire que distribués dans le cytoplasme environnant et formant ainsi un large complexe macromoléculaire [2–21]. On va ainsi identifier une protéine apparentée à la dystrophine comme la dystrobrévine avec 2 versions différentes (α et β) et 7 à 8 isoformes respectivement. Ensuite on va baptiser des protéines comme les 2 dystroglycanes (α et β), progressivement jusqu’à 6 sarcoglycanes (α, β, γ, δ, ε, ζ) puis 5 syntrophines différentes (α, β1, β2 et γ1, γ2). Des recherches sur les protéines associées permettront de découvrir la syncoïline, la desmusline (= synémine β) et la dysbindine. Les études sur la structure et la fonction de ces protéines allaient offrir aux chercheurs de multiples axes d’investigations. Ces protéines en interaction avec la dystrophine sont alors classées comme des glycoprotéines et/ou simplement des protéines associées. Un récent bilan actualisé pour mieux saisir la complexité de l’arrangement architectural autour de la membrane et du complexe entre dystrophine et ses multiples partenaires figure dans la récente étude citée en référence avec les principales voies de signalisation impliquées au sein du muscle cardiaque [22]. C’est cependant dans le muscle squelettique plus particulièrement au niveau des costamères [23] que l’on situa plusieurs types de connexions avec les laminines de la matrice extracellulaire. Parmi celles-ci figure une relation entre cavéoline-3 et dysferline d’une part [24] et cavéoline-3 et dystroglycane d’autre part [25] mais aussi un autre lien qui implique les intégrines [26] avec les laminines et le réseau d’actine sous-membranaire. Toutes ces protéines associées à la dystrophine sont indiquées dans le schéma présenté Fig. 1. Pour autant il fut rapidement évident que l’ensemble de ces protéines ne se trouvait pas toutes dans un même tissu ni dans un même compartiment cellulaire. Cela est illustré par exemple dans un travail original sur les syntrophines qui forment divers types de complexes macromoléculaires autour de la dystrophine selon qu’il s’agisse du muscle squelettique, de la jonction neuro-musculaire, du système nerveux central et/ou de la rétine [27] (Figure 1).
 |
Figure 1 Diagramme schématique permettant une vue d’ensemble du complexe macromoléculaire autour de la dystrophine et de ses partenaires directs et indirects. Au sein de la membrane du muscle squelettique s’ancrent le bêta-dystroglycane, la dysferline et les intégrines. Dans la matrice extracellulaire existe un réseau qui connecte entre elles ces protéines via l’alpha dystroglycane et/ou diverses laminines. Dans le cytoplasme on trouve des associations avec le réseau d’actine sous-membranaire via la dystrophine et diverses protéines associées. Ainsi dans ces 3 compartiments, matrice extracellulaire, cytoplasme et membrane, est présent un enchevêtrement complexe de protéines dont pour la plupart la découverte est en relation avec la dystrophine il y a 30 ans. |
Cependant, les connaissances acquises ne vont pas se limiter à la découverte de nouveaux partenaires mais aussi à la dystrophine elle-même. Ainsi l’épissage séquentiel du gène DMD indique que la longueur totale de l’ADNc est de 11, 3 kb avec 79 exons [28]. Rapidement il fut constaté que l’analyse du gène codant pour la dystrophine allait donner naissance à une grande famille de protéines dérivées [29]. Puis cette famille de protéines sera progressivement complétée par la découverte d’une protéine homologue et ubiquitaire l’utrophine [30] et de ses nombreuses isoformes [31, 32]. D’autres protéines apparentées viendront enrichir le tableau comme les dystrobrévines (plusieurs isoformes α et β), la DRP2 (Dystrophin-Related Protein 2) et la dystrotéline [33–35]. Cet ensemble finira par former non seulement « la super famille des dystrophines » [36] mais un large éventail de protéines relativement homologues avec un arbre phylogénétique complet comme le montre l’illustration présentée dans le travail original de Jin et al. [37]. Par ailleurs au cours de ces 30 années de recherche dans le domaine de la dystrophine, c’est l’environnement de ce complexe macromoléculaire qui sera mieux décrit comme le montre l’article de synthèse de Constantin [38]. On va en particulier indiquer les relations entre le complexe dystrophine d’une part avec les canaux ioniques PMCA, Nav1.5, TRPC1/4, mais aussi la cavéoline-3, la protéine nNOS, et d’autre part une abondante cascade de voies de signalisation comme cela est illustré dans ce document de synthèse de 2014 [38]. De plus des particularités sur les relations entre dystrophine et l’enveloppe nucléaire par exemple [39], mais également avec la participation de la myosine spécifique de type Myo18A [40] sont à prendre en compte dans cette architecture complexe autour de la dystrophine.
Conséquences d’une altération de la dystrophine
Que cela soit une absence totale ou une déficience partielle (protéine tronquée et/ou en quantité diminuée) en dystrophine, les conséquences se révèlent de plusieurs façons. Pour les muscles squelettiques il y a perte de l’intégrité membranaire des fibres musculaires responsable d’une dystrophie musculaire [41, 42]. Au niveau du muscle cardiaque l’expression anormale de la dystrophine est responsable d’une cardiomyopathie isolée ou le plus souvent associée à la dystrophie musculaire [43, 44]. Par ailleurs, il faut noter qu’il existe des cardiomyopathies liées à l’absence de dystrophine aussi bien chez les patients DMD et/ou BMD que chez les femmes dites transmettrices de ces pathologies.
Dans une moindre mesure, la déficience en dystrophine dans les muscles lisses peut générer des conséquences vasculaires et digestives [45–47] que l’on retrouve en partie chez les patients [48]. La dystrophine est également présente sous 5 isoformes différentes dans le système nerveux central et sous 2 isoformes dans la rétine. Les mutations du gène de la dystrophine sont responsables chez une majorité de patients de troubles cognitifs et/ou neuropsychiatriques plus ou moins sévères en fonction du nombre d’isoformes touchées [49]. Au niveau de la rétine, l’expression anormale de la dystrophine est essentiellement responsable d’une altération de l’électrorétinogramme chez les patients [50].
Ces défauts directement liés à une altération sur la séquence de la dystrophine (allant de l’absence à une perte partielle de séquence interne), va conduire à un écroulement plus ou moins grave de l’organisation des partenaires formant normalement le complexe macromoléculaire, au sens large, autour de la dystrophine. D’autre part, l’absence de dystrophine perturbe plusieurs voies de signalisation intracellulaire au niveau de la fibre musculaire squelettique avec pour conséquences un rôle néfaste des ions calcium, des espèces d’oxygène réactif (ROS) et de l’oxyde nitrique (NO) qui contribuent chacun au développement de la dystrophie musculaire [51].
Pathologies associées au complexe membranaire autour de la dystrophine
Un travail important présenté à la fin de l’année 2015, permet une mise à jour des altérations potentielles provoquées par une absence de la dystrophine. Illustrée par de nombreux schémas, cette revue résume les diverses données acquises par la recherche dans ce domaine. En particulier le schéma du portrait-robot de la dystrophine est repris pour y indiquer diverses relations majeures de voisinages avec les protéines environnantes [51].
Les protéines que l’on trouve en interaction directe et/ou indirecte autour de la dystrophine représentent un large complexe macromoléculaire dont beaucoup sont responsables de dystrophies musculaires isolées suite à l’identification de la dystrophine.
Cela concerne bien sûr l’association directe de l’altération de la dystrophine avec les pathologies de Duchenne et de Becker (DMD/BMD) mais bien audelà. Certains types de dystrophies dites congénitales (CMD) reliées à des altérations de la laminine α2 ou de l’alpha-dystroglycane ou également de l’intégrine alpha7 apparaissent en lien indirect avec la dystrophine. Il existe aussi un large éventail de dystrophies des ceintures de transmission autosomique récessive (LGMD2) également reliées au complexe macromoléculaire autour de la dystrophine. Un bilan actuel, inspiré par la revue proposée par Wicklund et Kissel [52], qui ne cesse de s’enrichir comme le montre une récente étude [53], conduit à illustrer l’état des lieux par un schéma récapitulatif présenté sur la figure 2.
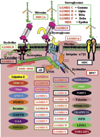 |
Figure 2 Spectre actuel des pathologies musculaires associées autour du complexe macromoléculaire entre la dystrophine et ses nombreux partenaires au sens large. Cliniquement si l’on avait déjà répertorié divers types de dystrophies musculaires, CMD, DMD, LGMD, aucune connaissance n’existait sur les protéines responsables avant l’identification de la dystrophine il y a 30 ans. Actuellement, et cela ne cesse d’évoluer en particulier dans le domaine des LGMD de type 2, une identification précise existe pour la plupart d’entre elles. |
Conclusion
La dystrophine est une protéine complexe et pleine de diversité : taille du gène, formes différentes suivant les tissus, nombreux partenaires, protéine de structure mais également « régulatrice » dans l’homéostasie de la fibre musculaire… Ces éléments expliquent au moins en partie l’absence de thérapeutique curative après 30 années de recherche. La particularité du tissu musculaire avec son propre système de régénération, sa densité et son volume de distribution est certainement l’autre contrainte majeure pour avancer sur la voie du traitement.
Malgré toutes ces difficultés les progrès sont indiscutables avec pour objectif réaliste actuel, l’amélioration du phénotype en restaurant une partie des fonctions de la dystrophine.
Les stratégies développées ont été, et doivent rester, multiples. Les thérapies cellulaires et géniques ont été sources de grand espoir au départ, avec de réels succès sur le plan expérimental, mais sans pouvoir apporter vraiment, actuellement, la preuve d’un résultat thérapeutique fonctionnel en clinique. Plus récemment des stratégies plus subtiles, saut d’exon thérapeutique et translecture de codon stop prématurés (mutations non-sens), ont amené un nouvel espoir avec des résultats fonctionnels modérés mais relativement significatifs chez les patients. Enfin différentes pharmacothérapies visant à atténuer les conséquences de l’absence de dystrophine au niveau du cœur et/ou du muscle squelettique ont pris progressivement un intérêt croissant. De nombreuses études et méta-analyses confirment actuellement l’efficacité des corticostéroïdes pour ralentir l’aggravation de la faiblesse musculaire que ce soit avant ou après la perte de la marche. L’efficacité des inhibiteurs de l’enzyme de conversion (IEC) sur le développement de la cardiomyopathie est également bien documentée [54]. D’autres thérapeutiques ou supplémentations diététiques font encore l’objet de recherche [55–61].
Il semble donc que l’on s’achemine vers des thérapeutiques plurielles qui pourraient être associées, de manière plus ou moins personnalisée, en fonction des données génétiques et cliniques. Bref une gestion multi-thérapeutique de l’absence de dystrophine, un peu à l’image de la prise en charge multidisciplinaire, apparaît actuellement indispensable pour offrir le meilleur traitement au patient atteint de la dystrophie musculaire de Duchenne.
Interview en 6 questions du Pr Michel Koenig
-
À la «Gordon conférence» en 1987 vous présentiez, hors programme, le séquençage complet de la Dystrophine, quel souvenir en avez-vous ?
C’était en fait au congrès de l'American Society of Human Genetics en octobre 1987 à San Diego. Suite à ma présentation sur la découverte de la structure et des domaines de la dystrophine et des points chauds de délétion du gène DMD, j’ai eu le prix de présentation orale catégorie post-doctorant-recherche fondamentale.
-
Puis les partenaires de la Dystrophine devinrent de plus en plus nombreux durant les années suivantes, lequel fut pour vous le plus surprenant ?
Le plus symbolique a été la découverte du groupe de Kevin Campbell (chez qui mon étudiant Franck Duclos est allé bien plus tard) du complexe dystroglycane/sarcoglycane et l’association avec la laminine α2 et le collagène VI, tous directement ou indirectement impliqués dans des dystrophies musculaires.
-
Un total d’environ 2 000 kb avec un grand gène, finalement une séquence de11, 3 kbdu cDNA et79 exons pour la partie codante correspondant à la Dystrophine mais aussi de très grands introns, pensez-vous qu’il s’y cache encore des surprises ?
Le mécanisme d’épissage de ces grands introns, le lien avec la présence des nombreux sites d’initiation de la transcription et les points chauds de délétion n’ont pas encore été élucidés. J’ai appris récemment (par Sylvie Tuffery) qu’il existe un exon 2a alternatif fréquemment utilisé dans le très grand intron 1, et dont le seul rôle apparent est d’être abortif ! Curieux, non ?
-
L’existence de nombreux promoteurs alternatifs fut-elle une surprise pour vous ?
Absolument, et c’est certainement aussi une part du mystère. Je suis en train de découvrir qu’il existe le même phénomène dans le gène SYNE1 (nesprine-1) de la même famille des spectrine-repeats et qui est muté dans une ataxie héréditaire (ma nouvelle vie). La spectrine bêta 2 est aussi mutée dans une ataxie héréditaire.
-
Après la découverte de la Dystrophine, avez-vous soupçonné l’existence d’une protéine homologue l’Utrophine ?
Non, pas du tout. En fait, il y a 2 protéines homologues (DRP1 = utrophine et DRP2) plus les nesprines, la dystonine, sans oublier les spectrines, les alpha-actinines, les filamines… une longue histoire.
-
Quel serait votre « préférence » pour traiter la dystrophie de Duchenne, restaurer la Dystrophine manquante ou favoriser la surexpression de l’Utrophine ?
Je vais citer (approximativement) une phrase du Pr Arnold Munnich, dans son dernier livre : « on voudrait prêter trop de pouvoir aux médecins ».
Liens d’intérêt
L’auteur déclare n’avoir aucun lien d’intérêt concernant les données publiées dans cet article.
Références
- Koenig M, Hoffman EP, Bertelson CJ, et al. Complete cloning of the Duchenne muscular dystrophy (DMD) cDNA and preliminary genomic organization of the DMD gene in normal and affected individuals. Cell 1987; 50 : 509–17. [CrossRef] [PubMed] [Google Scholar]
- Campbell KP, Kahl SD. Association of dystrophin and an integral membrane glycoprotein. Nature 1989; 338 : 259–62. [CrossRef] [PubMed] [Google Scholar]
- Carr C, Fischbach GD, Cohen JB. A novel 87, 000-Mr protein associated with acetylcholine receptors in Torpedo electric organ and vertebrate skeletal muscle. J Cell Biol 1989; 109 : 1753–64. [CrossRef] [PubMed] [Google Scholar]
- Fisher LW, Termine JD, Young MF. Deduced protein sequence of bone small proteoglycan I (biglycan) shows homology with proteoglycan II (decorin) and several nonconnective tissue proteins in a variety of species. J Biol Chem 1989; 264 : 4571–6. [PubMed] [Google Scholar]
- Ibraghimov-Beskrovnaya O, Ervasti JM, Leveille CJ, et al. Primary structure of dystrophin-associated glycoproteins linking dystrophin to the extracellular matrix. Nature 1992; 355 : 696–702. [CrossRef] [PubMed] [Google Scholar]
- Fardeau M, Matsumura K, Tomé FM, et al. Deficiency of the 50 kDa dystrophin associated glycoprotein (adhalin) in severe autosomal recessive muscular dystrophies in children native from European countries. CR Acad Sci III 1993; 316 : 799–804. [Google Scholar]
- Gee SH, Blacher RW, Douville PJ, et al. Laminin-binding protein 120 from brain is closely related to the dystrophin-associated glycoprotein, dystroglycan, and binds with high affinity to the major heparin binding domain of laminin. J Biol Chem 1993; 268 : 14972–80. [PubMed] [Google Scholar]
- Yang B, Ibraghimov-Beskrovnaya O, Moomaw CR, et al. Heterogeneity of the 59-kDa dystrophin-associated protein revealed by cDNA cloning and expression. JBiol Chem 1994; 269 : 6040–4. [Google Scholar]
- Peters MF, Kramarcy NR, Sealock R, Froehner SC. Beta 2-Syntrophin: localization at the neuromuscular junction in skeletal muscle. Neuroreport 1994; 5 : 1577–80. [CrossRef] [PubMed] [Google Scholar]
- Lim LE, Duclos F, Broux O, et al. Beta-sarcoglycan: characterization and role in limb-girdle muscular dystrophy linked to 4q12. Nat Genet 1995; 11 : 257–65. [CrossRef] [PubMed] [Google Scholar]
- Song KS, Scherer PE, Tang Z, et al. Expression of caveolin-3 in skeletal, cardiac, and smooth muscle cells. Caveolin-3 is a component of the sarcolemma and co-fractionates with dystrophin and dystrophin-associated glycoproteins. JBiol Chem 1996; 271 : 15160–5. [CrossRef] [Google Scholar]
- Jung D, Leturcq F, Sunada Y, et al. Absence of gamma-sarcoglycan (35 DAG) in autosomal recessive muscular dystrophy linked to chromosome 13q12. FEBS Lett 1996; 381 : 15–20. [PubMed] [Google Scholar]
- Jung D, Duclos F, Apostol B, et al. Characterization of deltasarcoglycan, a novel component of the oligomeric sarcoglycan complex involved in limb-girdle muscular dystrophy. J Biol Chem 1996; 271 : 32321–9. [CrossRef] [PubMed] [Google Scholar]
- Straub V, Ettinger AJ, Durbeej M, et al. Epsilon-sarcoglycan replaces alpha-sarcoglycan in smooth muscle to form a unique dystrophin-glycoprotein complex. J Biol Chem 1999; 274 : 27989–96. [CrossRef] [PubMed] [Google Scholar]
- Crosbie RH, Heighway J, Venzke DP, et al. Sarcospan, the 25-kDa transmembrane component of the dystrophin-glycoprotein complex. J Biol Chem 1997; 272 : 31221–4. [CrossRef] [PubMed] [Google Scholar]
- Piluso G, Mirabella M, Ricci E, et al. Gamma1- and gamma2-syntrophins, two novel dystrophin-binding proteins localized in neuronal cells. J Biol Chem 2000; 275 : 15851–60. [CrossRef] [PubMed] [Google Scholar]
- Mizuno Y, Thompson TG, Guyon JR, et al. Desmuslin, an intermediate filament protein that interacts with alpha -dystrobrevin and desmin. Proc Natl Acad Sci USA 2001; 98 : 6156–61. [CrossRef] [Google Scholar]
- Newey SE, Howman EV, Ponting CP, et al. Syncoilin, a novel member of the intermediate filament superfamily that interacts with alpha-dystrobrevin in skeletal muscle. J Biol Chem 2001; 276 : 6645–55. [CrossRef] [PubMed] [Google Scholar]
- Benson MA, Newey SE, Martin-Rendon E, et al. Dysbindin, a novel coiled-coil-containing protein that interacts with the dystrobrevins in muscle and brain. J Biol Chem 2001; 276: 24232–41. [CrossRef] [PubMed] [Google Scholar]
- Wheeler MT, Zarnegar S, McNally EM. Zeta-sarcoglycan, a novel component of the sarcoglycan complex, is reduced in muscular dystrophy. Hum Mol Genet 2002; 11 : 2147–54. [CrossRef] [PubMed] [Google Scholar]
- Shiga K, Yoshioka H, Matsumiya T, et al. Zeta-sarcoglycan is a functional homologue of gamma-sarcoglycan in the formation of the sarcoglycan complex. Exp Cell Res 2006; 312 : 2083–92. [PubMed] [Google Scholar]
- Taghli-Lamallem O, Jagla K, Chamberlain JS, et al. Mechanical and non-mechanical functions of Dystrophin can prevent cardiac abnormalities in Drosophila. Exp Gerontol 2014; 49 : 26–34. [CrossRef] [PubMed] [Google Scholar]
- Anastasi G, Cutroneo G, Santoro G. Costameric proteins in human skeletal muscle during muscular inactivity. J Anat 2008; 213 : 284–95. [CrossRef] [PubMed] [Google Scholar]
- Matsuda C, Hayashi YK, Ogawa M, et al. The sarcolemmal proteins dysferlin and caveolin-3 interact in skeletal muscle. Hum Mol Genet 2001; 10 : 1761–6. [CrossRef] [PubMed] [Google Scholar]
- Sotgia F, Lee JK, Das K, et al. Caveolin-3 directly interacts with the C-terminal tail of beta -dystroglycan. Identification of a central WW-like domain within caveolin family members. J Biol Chem 2000; 275 : 38048–58. [CrossRef] [PubMed] [Google Scholar]
- Paul AC, Sheard PW, Kaufman SJ, Duxson MJ. Localization of alpha 7 integrins and dystrophin suggests potential for both lateral and longitudinal transmission of tension in large mammalian muscles. Cell Tissue Res 2002; 308 : 255–65. [CrossRef] [PubMed] [Google Scholar]
- Pilgram GS, Potikanond S, Baines RA, et al. The roles of the dystrophin-associated glycoprotein complex at the synapse. Mol NeuroBiol 2010; 41 : 1–21. [PubMed] [Google Scholar]
- Bougé AL, Murauer E, Beyne E, et al. Targeted RNA-Seq profiling of splicing pattern in the DMD gene: exons are mostly constitutively spliced in human skeletal muscle. Sci Rep 2017; 7 : 39094. [CrossRef] [PubMed] [Google Scholar]
- Sadoulet-Puccio HM, Kunkel LM. Dystrophin and its isoforms. Brain Pathol 1996; 6 : 25–35. [PubMed] [Google Scholar]
- Khurana TS, Watkins SC, Kunkel LM. The subcellular distribution of chromosome 6-encoded dystrophin-related protein in the brain. J Cell Biol 1992; 119 : 357–66. [CrossRef] [PubMed] [Google Scholar]
- Jimenez-Mallebrera C, Davies K, Putt W, Edwards YH. A study of short utrophin isoforms in mice deficient for full-length utrophin. Mamm Genome 2003; 14 : 47–60. [CrossRef] [PubMed] [Google Scholar]
- Zuellig RA, Bornhauser BC, Knuesel I, et al. Identification and characterisation of transcript and protein of a new short N-terminal utrophin isoform. J Cell Biochem 2000; 77 : 418–31. [CrossRef] [PubMed] [Google Scholar]
- Roberts RG, Freeman TC, Kendall E, et al. Characterization of DRP2, a novel human dystrophin homologue. Nat Genet 1996; 13 : 223–6. [CrossRef] [PubMed] [Google Scholar]
- Blake DJ, Nawrotzki R, Peters MF, et al. Isoform diversity of dystrobrevin, the murine 87-kDa postsynaptic protein. J Biol Chem 1996; 271 : 7802–10. [CrossRef] [PubMed] [Google Scholar]
- Bohm SV, Roberts RG. Expression of members of the dystrophin, dystrobrevin, and dystrotelin superfamily. Crit Rev Eukaryot Gene Expr 2009; 19 : 89–108. [PubMed] [Google Scholar]
- Fabbrizio E, Pons F, Robert A, et al. The dystrophin superfamily: variability and complexity. J Musc Res Cell Motil 1994; 15: 595–606. [CrossRef] [Google Scholar]
- Jin H, Tan S, Hermanowski J, Bohm S, et al. The dystrotelin, dystrophin and dystrobrevin superfamily: new paralogues and old isoforms. BMC Genomics 2007; 8 : 19. [CrossRef] [PubMed] [Google Scholar]
- Constantin B. Dystrophin complex functions as a scaffold for signalling proteins. Biochim Biophys Acta 2014; 1838 : 635–42. [PubMed] [Google Scholar]
- Iyer SR, Shah SB, Valencia AP, et al. Altered nuclear dynamics in MDX myofibers. J Appl Physiol (1985) 2017; 122 : 470–81. [CrossRef] [PubMed] [Google Scholar]
- Cao JM, Cheng XN, Li SQ, et al. Identification of novel MYO18A interaction partners required for myoblast adhesion and muscle integrity. Sci Rep 2016; 6 : 36768. [CrossRef] [PubMed] [Google Scholar]
- Rahimov F, Kunkel LM. The cell biology of disease: cellular and molecular mechanisms underlying muscular dystrophy. J Cell Biol 2013; 201: 499–510. [CrossRef] [PubMed] [Google Scholar]
- Serrano AL, Munoz-Canoves P. Fibrosis development in early-onset muscular dystrophies: mechanisms and translational implications. Semin Cell Dev Biol 2017; 64: 181–90. [CrossRef] [PubMed] [Google Scholar]
- Nakamura A. X-linked dilated cardiomyopathy: a cardiospecific phenotype of dystrophinopathy. Pharmaceuticals (Basel) 2015; 8 : 303–20. [PubMed] [Google Scholar]
- Cripe LH, Tobias JD. Cardiac considerations in the operative management of the patient with Duchenne or Becker muscular dystrophy. Paediatr Anaesth 2013; 23 : 777–84. [CrossRef] [PubMed] [Google Scholar]
- Rauch U, Shami A, Zhang F, et al. Increased neointimal thickening in dystrophin-deficient mdx mice. PLoS One 2012; 7: e29904. [PubMed] [Google Scholar]
- Latroche C, Matot B, Martins-Bach A, et al. Structural and functional alterations of skeletal muscle microvasculature in dystrophin-deficient mdx mice. Am J Pathol 2015; 185 : 2482–94. [PubMed] [Google Scholar]
- Manning J, Buckley MM, O’Halloran KD, O’Malley D. In vivo neutralization of IL-6 receptors ameliorates gastrointestinal dysfunction in dystrophin-deficient mdx mice. Neurogastroenterol Motil 2016;28: 1016–26. [PubMed] [Google Scholar]
- Lo Cascio CM, Goetze O, Latshang TD, et al. Gastrointestinal dysfunction in patients with Duchenne muscular dystrophy. PLoS One 2016;11:e0163779. [PubMed] [Google Scholar]
- Anand A, Tyagi R, Mohanty M, et al. Dystrophin induced cognitive impairment: mechanisms, models and therapeutic strategies. Ann Neurosci 2015; 22 : 108–18. [CrossRef] [PubMed] [Google Scholar]
- Barboni MT, Martins CM, Nagy BV, et al. Dystrophin Is required for proper functioning of luminance and red-green cone opponent mechanisms in the human retina. Invest OphthalMol Vis Sci 2016; 57 : 3581–7. [CrossRef] [PubMed] [Google Scholar]
- Allen DG, Whitehead NP, Froehner SC. Absence of dystrophin disrupts skeletal muscle signaling : roles of Ca2+, reactive oxygen species, and nitric oxide in the development of muscular dystrophy. Physiol Rev 2016; 96: 253–305. [Google Scholar]
- Wicklund MP, Kissel JT. The limb-girdle muscular dystrophies. Neurol Clin 2014; 32 : 729–49. [CrossRef] [PubMed] [Google Scholar]
- Ghaoui R, Benavides T, Lek M, et al. TOR1AIP1 as a cause of cardiac failure and recessive limb-girdle muscular dystrophy. Neuromuscul Disord 2016; 26 : 500–3. [PubMed] [Google Scholar]
- El-Aloul B, Altamirano-Diaz L, Zapata-Aldana E, et al. Pharmacological therapy for the prevention and management of cardiomyopathy in Duchenne muscular dystrophy: a systematic review. Neuromuscul Disord 2017; 27 : 4–14. [PubMed] [Google Scholar]
- Rybalka E, Timpani CA, Stathis CG, et al. Metabogenic and nutriceutical approaches to address energy dysregulation and skeletal muscle wasting in Duchenne muscular dystrophy. Nutrients 2015; 7:9734–67. [CrossRef] [PubMed] [Google Scholar]
- Ballmann C, Denney TS, Beyers RJ, et al. Lifelong quercetin enrichment and cardioprotection in Mdx/Utrn+/- mice. Am J Physiol Heart Circ Physiol 2017; 312 : H128–40. [PubMed] [Google Scholar]
- Spaulding HR, Ballmann CG, Quindry JC, Selsby JT. Longterm quercetin dietary enrichment partially protects dystrophic skeletal muscle. PLoS One 2016; 11 : e0168293 [PubMed] [Google Scholar]
- Miyatake S, Shimizu-Motohashi Y, Takeda S, Aoki Y. Antiinflammatory drugs for Duchenne muscular dystrophy: focus on skeletal muscle-releasing factors. DrugDes Devel Ther 2016; 10 : 2745–58. [CrossRef] [Google Scholar]
- Reinig AM, Mirzaei S, Berlau DJ. Advances in the treatment of Duchenne muscular dystrophy: new and emerging pharmacotherapies. Pharmacotherapy 2017; 37 : 492–9. [PubMed] [Google Scholar]
- Zhou J, Xin J, Niu Y, Wu S. DMDtoolkit: a tool for visualizing the mutated dystrophin protein and predicting the clinical severity in DMD. BMC Bioinformatics 2017; 18 : 87–97. [CrossRef] [PubMed] [Google Scholar]
- Goemans N, Tulinius M, Kroksmark AK, et al. Comparison of ambulatory capacity and disease progression of Duchenne muscular dystrophy subjects enrolled in the drisapersen DMD114673 study with a matched natural history cohort of subjects on daily corticosteroids. Neuromuscul Disord 2017; 27 : 203–13. [PubMed] [Google Scholar]
Liste des figures
 |
Figure 1 Diagramme schématique permettant une vue d’ensemble du complexe macromoléculaire autour de la dystrophine et de ses partenaires directs et indirects. Au sein de la membrane du muscle squelettique s’ancrent le bêta-dystroglycane, la dysferline et les intégrines. Dans la matrice extracellulaire existe un réseau qui connecte entre elles ces protéines via l’alpha dystroglycane et/ou diverses laminines. Dans le cytoplasme on trouve des associations avec le réseau d’actine sous-membranaire via la dystrophine et diverses protéines associées. Ainsi dans ces 3 compartiments, matrice extracellulaire, cytoplasme et membrane, est présent un enchevêtrement complexe de protéines dont pour la plupart la découverte est en relation avec la dystrophine il y a 30 ans. |
| Dans le texte | |
 |
Figure 2 Spectre actuel des pathologies musculaires associées autour du complexe macromoléculaire entre la dystrophine et ses nombreux partenaires au sens large. Cliniquement si l’on avait déjà répertorié divers types de dystrophies musculaires, CMD, DMD, LGMD, aucune connaissance n’existait sur les protéines responsables avant l’identification de la dystrophine il y a 30 ans. Actuellement, et cela ne cesse d’évoluer en particulier dans le domaine des LGMD de type 2, une identification précise existe pour la plupart d’entre elles. |
| Dans le texte | |
Les statistiques affichées correspondent au cumul d'une part des vues des résumés de l'article et d'autre part des vues et téléchargements de l'article plein-texte (PDF, Full-HTML, ePub... selon les formats disponibles) sur la platefome Vision4Press.
Les statistiques sont disponibles avec un délai de 48 à 96 heures et sont mises à jour quotidiennement en semaine.
Le chargement des statistiques peut être long.


